糖鎖データベースからの目的単糖を含む多糖の検索と可視化
はじめに
digzymeにてインターン勤務させていただいております、東京大学修士2年の高栁龍です。大学では、タンパク質のリン酸化やタンパク質立体構造に関連した研究などを行っています。今回のtechblogでは、研究開発業務の一環として新たに開発しました、目的単糖を含む多糖の網羅的検索と可視化ツールであるGlycoSearcherを紹介いたします。
近年、デンプンや食物繊維で代表されるように、糖鎖など多糖の研究と産業利用が活発となってきています。新規の糖質を開発する需要が上がってきており、その中で多糖は構造の多様性が非常に高いものとして注目されています。そこで、様々な多糖を網羅的に検索するためのツールとして、新たにGlycoSearcherを開発いたしました。
多糖の記述方式とデータベース
今回対象としている糖化合物は、既に数十万を超える数が報告されており、データベース化されています。こうした多くの糖化合物の中から目的に沿った多糖を厳選し、合成経路の探索や酵素開発などの応用につなげるには、計算処理しやすい記述方式と網羅性の高いデータベースが必要となります。
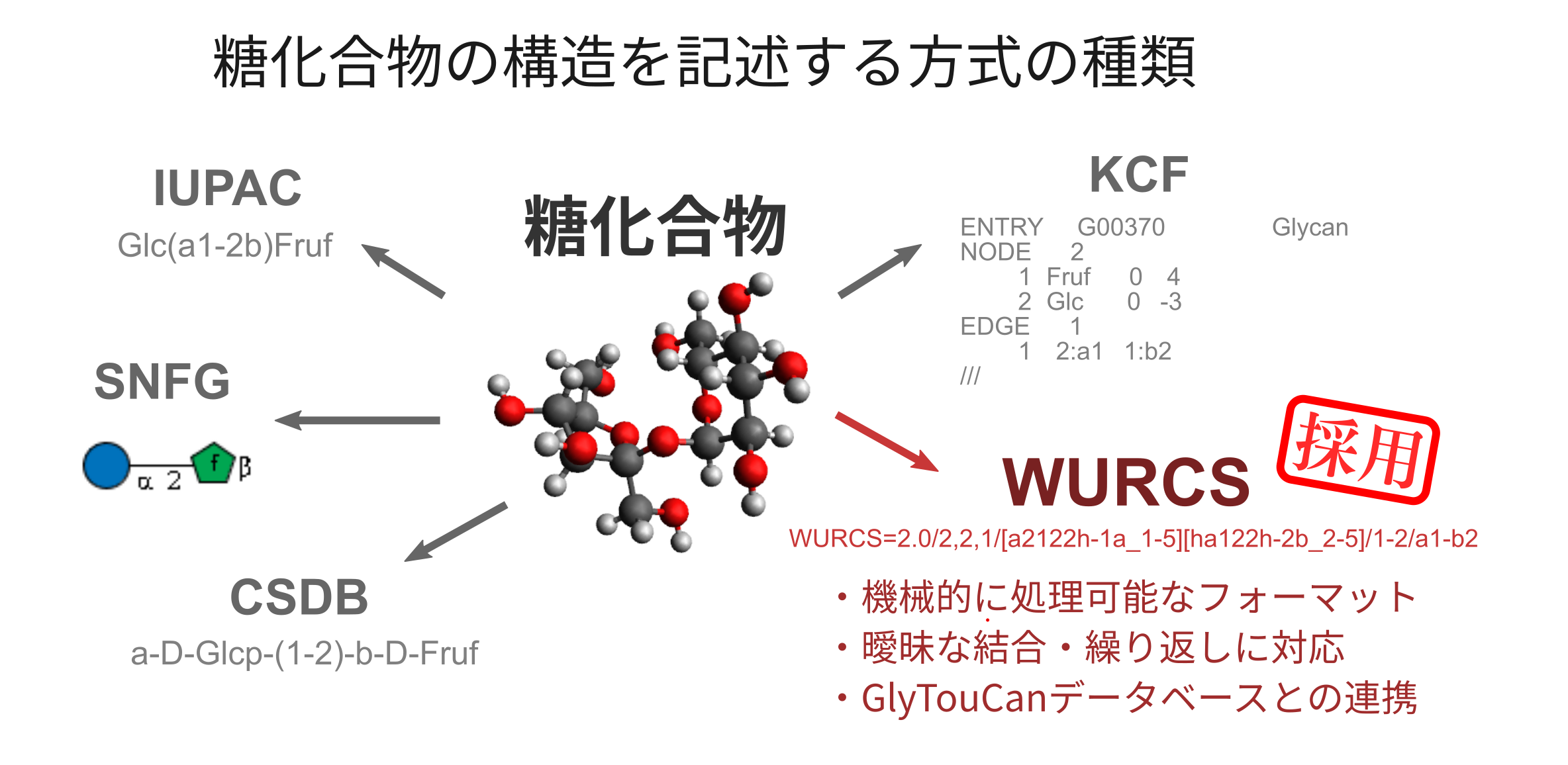
糖化合物の構造を記述する方式には様々なものが知られています(図1)。SNFGやKCFといった形式は可視化にすぐれていますが、構造情報の抽出や比較など応用的な計算処理には不向きです。一方、IUPACは、人と機械の両方が読み取り可能で簡潔な構造表現ですが、繰り返しなど複雑で曖昧な表現に対応するのは難しいです[1]。そこで、GlycoSearcherでは、計算処理に適し、繰り返し表現可能なWURCS形式、およびWURCS形式での糖化合物情報を収集したGlyTouCanデータベース[2]を採用しました。
(図1)
GlycoSearcherを用いた検索
ここにGlycoSearcherでは、膨大な量の候補から目的に沿った多糖を抽出することが可能です。例えば、グルコースやガラクトースなど、特定の単糖単位を含む多糖を検索することができます。さらに、多糖を構成する単糖単位を一部のものに限定する、フィルタリング機能も兼ね備えています。これにより、ある特定の単糖を原料とし、その他特定の糖のみを用いて合成可能な多糖を列挙することができます。
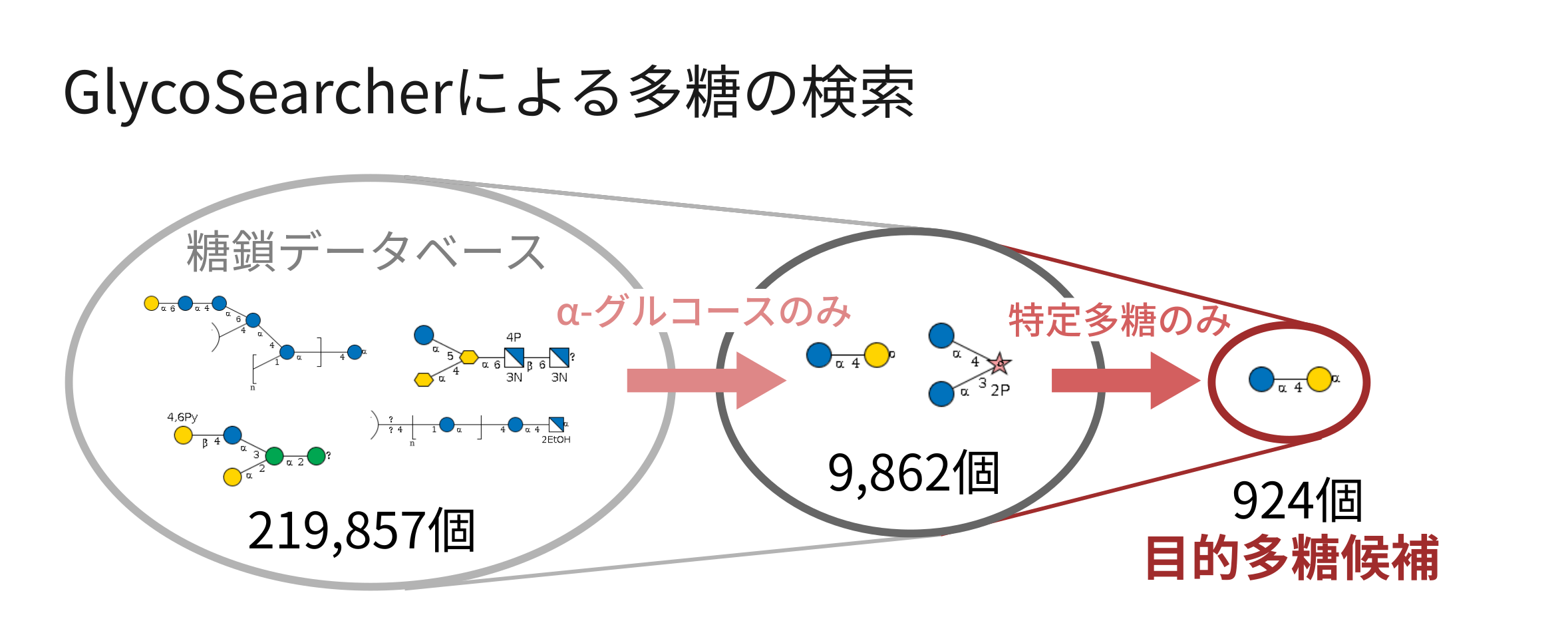
以下に、α-グルコースを例に実行した結果を紹介します(図2)。219,857個ある糖鎖の中から、α-グルコースを含む多糖を検索したところ、9,862個が検出されました。さらに、構成単糖をグルコース・ガラクトース・フルクトースのみに絞ったところ、924個まで候補が減少しました。
(図2)
多糖構造の可視化と特徴抽出
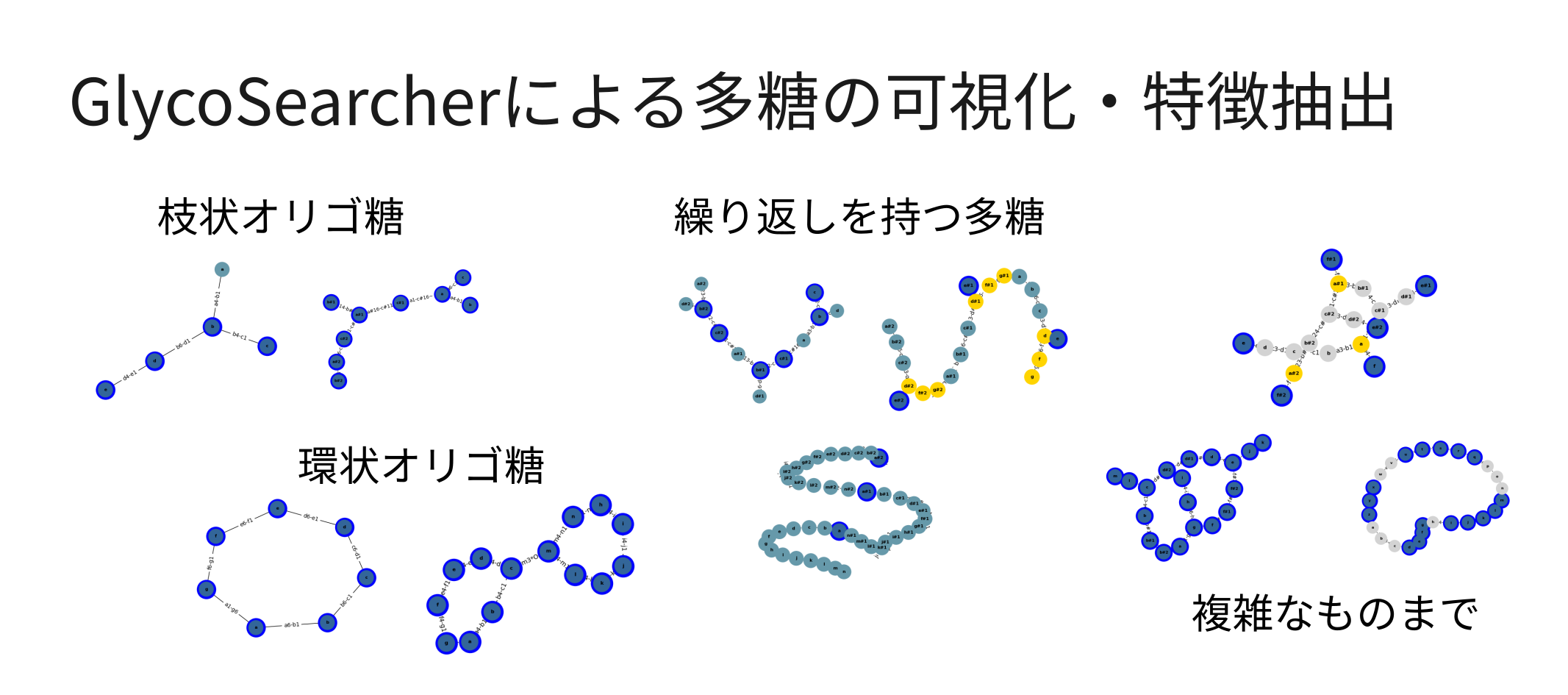
得られた検索結果は、効果的に可視化され、続く応用処理へと活かすことができます(図3)。WURCS形式の多糖をグラフとして再構成することで、数千個の検索結果を数分のうちに描きだすなど、高速な可視化が可能です。また、曖昧な繰り返し回数を持つ構造についても、特定の回数だけ繰り返しを展開することで、実質的な構造を可視化するのみならず、曖昧なままでは難しい構造間の比較など、更なる計算処理へとスムーズに移行させることができます。
検索結果の多糖がグラフ化されていることから、多糖構造に対する特徴抽出も可能です。例えば、得られた多糖構造の末端にグルコース単位が存在しているか、あるいは特定の構造(モチーフ)を含むか、といった計算を行うことができます。さらに、検索によってヒットした多糖について、PubChem[3]などの各種データベースと統合させることで、その一般名や関連する酵素情報と統合させることができ、多糖を含む反応についての情報を得ることが可能です。
(図3)
終わりに
今回開発したGlycoSearcherでは、目的の多糖をデータベースから網羅的に検索し、更なる計算処理へと応用させることができました。さらに、得られた目的多糖候補から情報を抽出しその合成に関与すると予測される酵素情報を取得することで、その後の酵素デザインのワークフローにつなげる体制を整えられました。
謝辞
糖化合物に関する知識の習得を始めとしたGlycoSearcherの開発には、事業開発部の礒崎さんに大変お世話になりました。この場を借りて感謝申し上げます。
参考文献
[1] 細田 正恵, 木下 聖子.「糖鎖関連インフォマティクスへの入り口」 JSBi Bioinformatics Review, 2(1), 87-95 (2021).
[2] https://glytoucan.org/
[3] https://pubchem.ncbi.nlm.nih.gov/
人工的な合成経路探索
はじめに
事業開発部の礒崎です。弊社では酵素反応による原料から目的物までの人工的な合成ルート探索を行っています。目的物と原料の化合物構造データを入力するだけで、原料から目的物を合成する可能性のあるルート候補を出力します。本ブログでは、その具体例として高強度ポリマーの原材料となる化合物4-アミノ桂皮酸をグルコースから合成するルートを予測し、反応を担う酵素を予測した結果をご紹介します。
合成経路探索に使用した材料
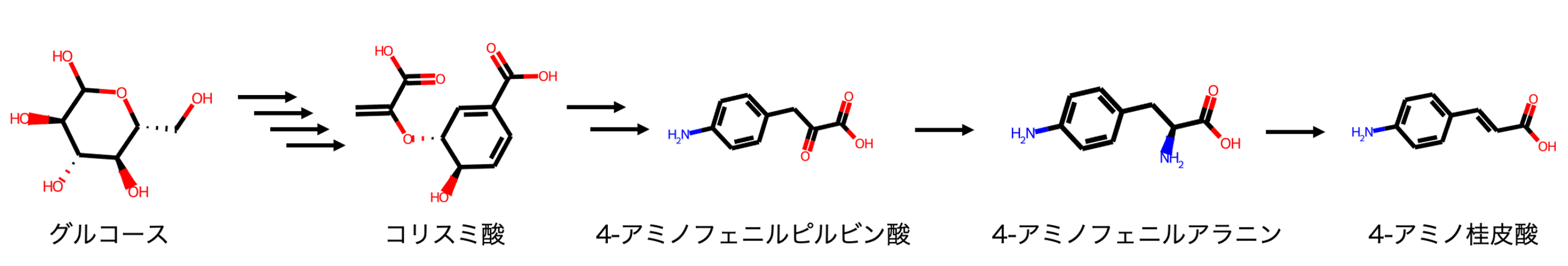
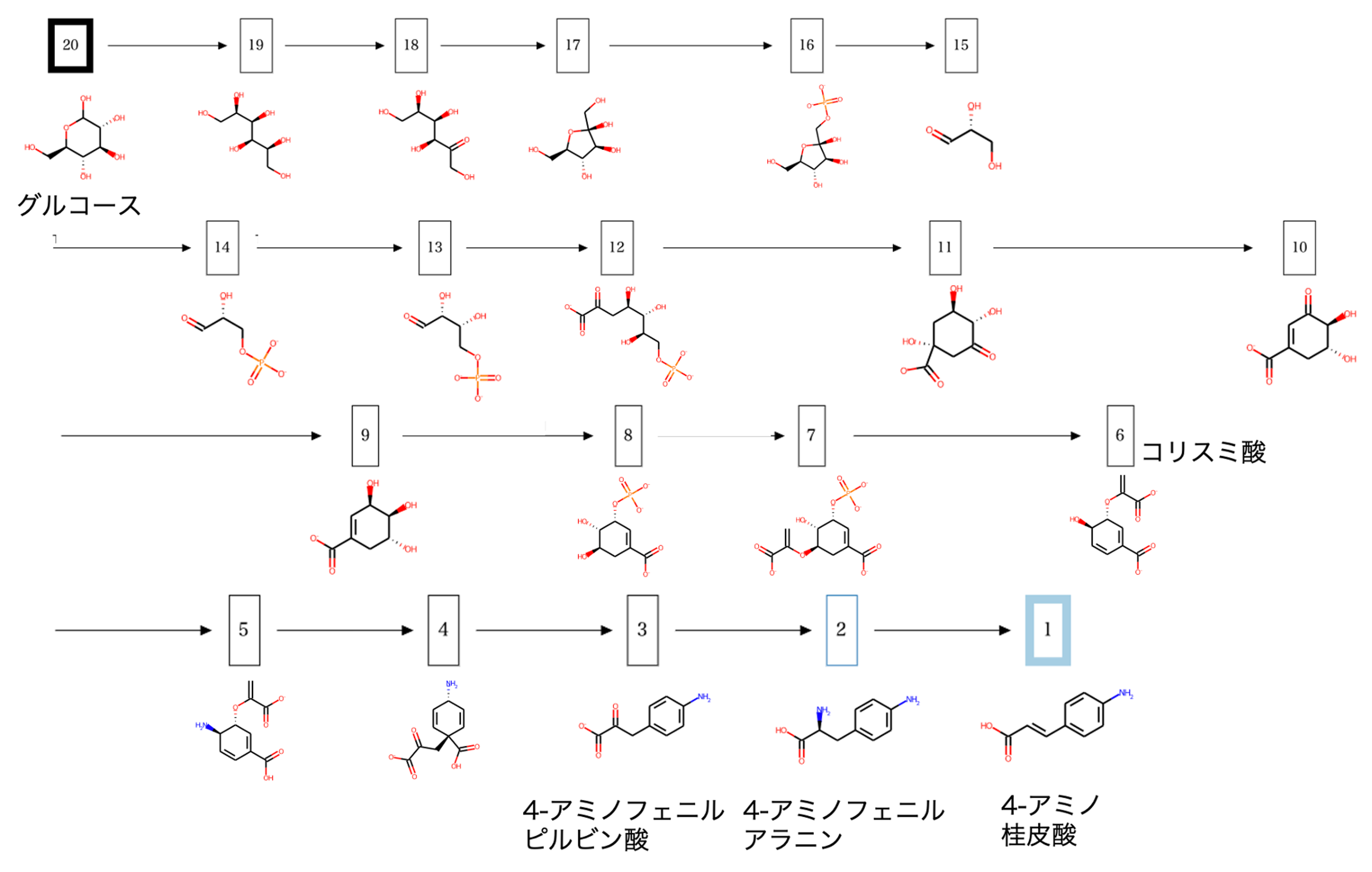
Tateyama et al., 2016において、高強度のポリマーを生産するための原材料として4-アミノ桂皮酸を使用しています。この4-アミノ桂皮酸を合成するために使用された経路が図1です。グルコースを原料としてStreptomyces venezuelae 由来のAminodeoxychorismate synthase (PapA)とS. pristinaespiralis由来の Aminodeoxychorismate synthase (PapBC)を導入した大腸菌により4-アミノフェニルアラニンを生産させます。さらに、この4-アミノフェニルアラニンを原料としてRhodotorula glutinisのPhenylalanine ammonia-lyase (RgPAL)を導入した大腸菌に加えて、4-アミノ桂皮酸を生産させます。
結果
1. 生合成経路探索
原料をグルコース、生成物を4-アミノ桂皮酸として入力することで図1のような人工合成経路が出力されました。グルコースからコリスミ酸の既知合成経路と同一の経路が出力され、4−アミノフェニルアラニンを介して4-アミノ桂皮酸を合成する経路が出力されました。
2. 類似反応探索
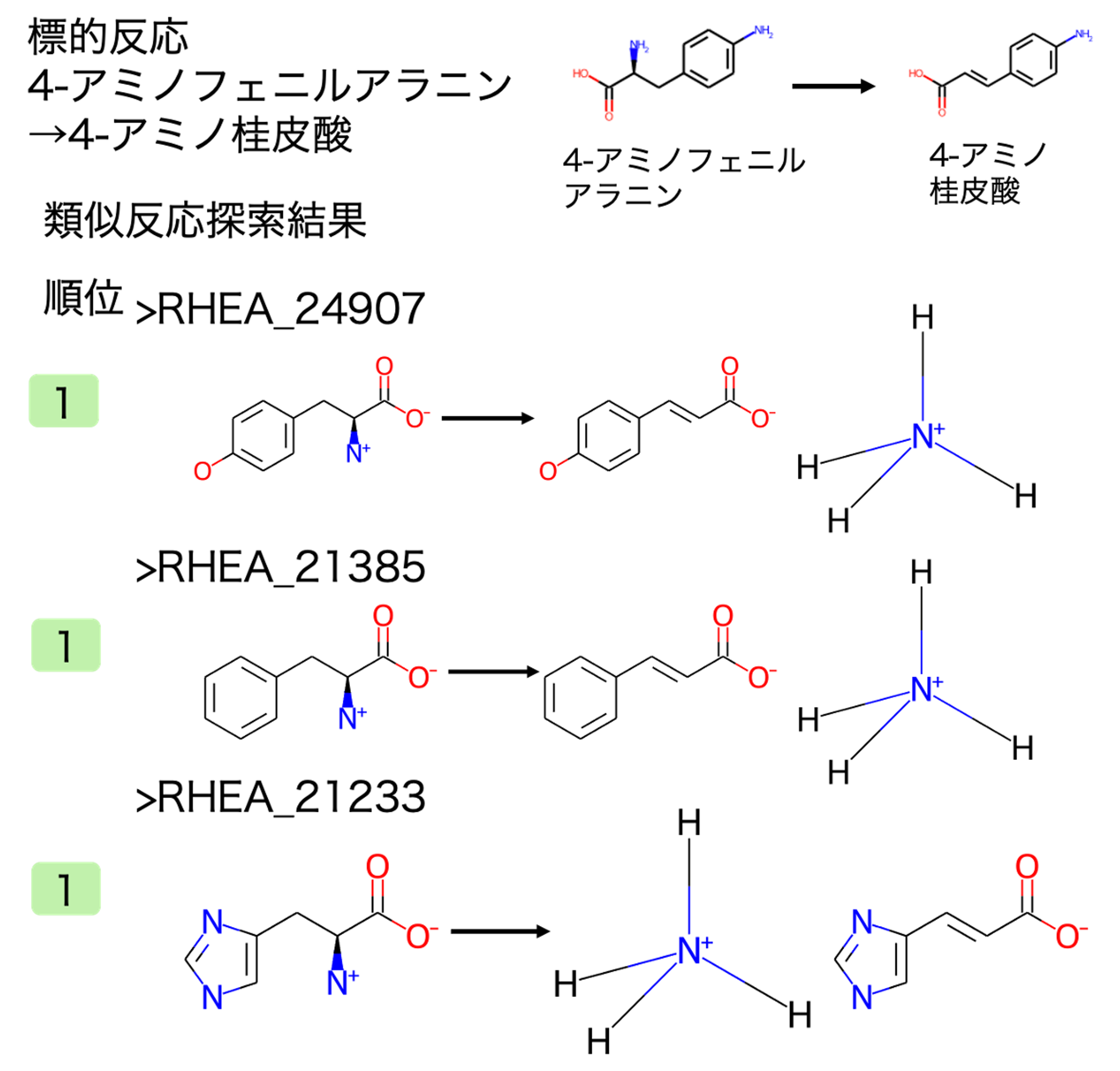
結果1で見出した人工的な合成経路のうち、4-アミノフェニルアラニン→4-アミノ桂皮酸の類似反応を探索しました。
類似反応探索により、アミノ基を脱離し、二重結合を生成する反応が抽出されました。標的反応との反応類似度が高い類似反応の一部とその順位を図2に示しました。標的反応と完全に一致する反応を含む類似反応が抽出されました。
3. 類似反応該当酵素探索
結果2で標的反応の類似反応を抽出しました。この類似反応を担う酵素配列をtaxonごとに抽出しました。絞り込んだ配列と論文中で使用された酵素を比較しました。Rhodotorula属、Eukaryotaドメイン、全taxonの3段階で配列を抽出しました(表1)。抽出した配列には、論文で使用された配列と90%以上の配列相同性を示すものが含まれていました。

終わりに
本ブログでは人工的な合成経路の探索を実演しました。材料として高強度ポリマーの原材料となる化合物4-アミノ桂皮酸をグルコース から合成する人工的な経路を探索しました。この経路のうち、4-アミノフェニルアラニンから4-アミノ桂皮酸を合成する酵素を類似反応酵素探索技術を用いて見つけることができるか試しました。上記の反応に対して、任意のtaxonごとに配列を抽出し、それぞれの配列数を示しました。実際に論文で使っていた酵素に非常に近い配列を含む複数の配列を抽出することができました。
謝辞
今回の合成経路探索には以下の論文のデータを利用させていただきました。
Tateyama et al., (2016) Ultrastrong, Transparent Polytruxillamides Derived from Microbial Photodimers. Maclomolecules.
