構造予測とMDシミュレーションを用いた酵素活性予測の実用例
はじめに
事業開発部の礒崎です。弊社では有用酵素探索の1つとして、分子動力学シミュレーションを用いた酵素活性予測を行っています。構造未知の酵素配列からその構造を予測し、標的化合物との複合体を分子動力学シミュレーションへ供します。その結果から、digzyme独自のスコアを算出し酵素活性を予測します。本ブログでは、その具体例としてthiolaseの1つoleAという酵素の類似配列から実際に活性をもつものを予測した結果をご紹介します。
酵素活性予測に使った材料
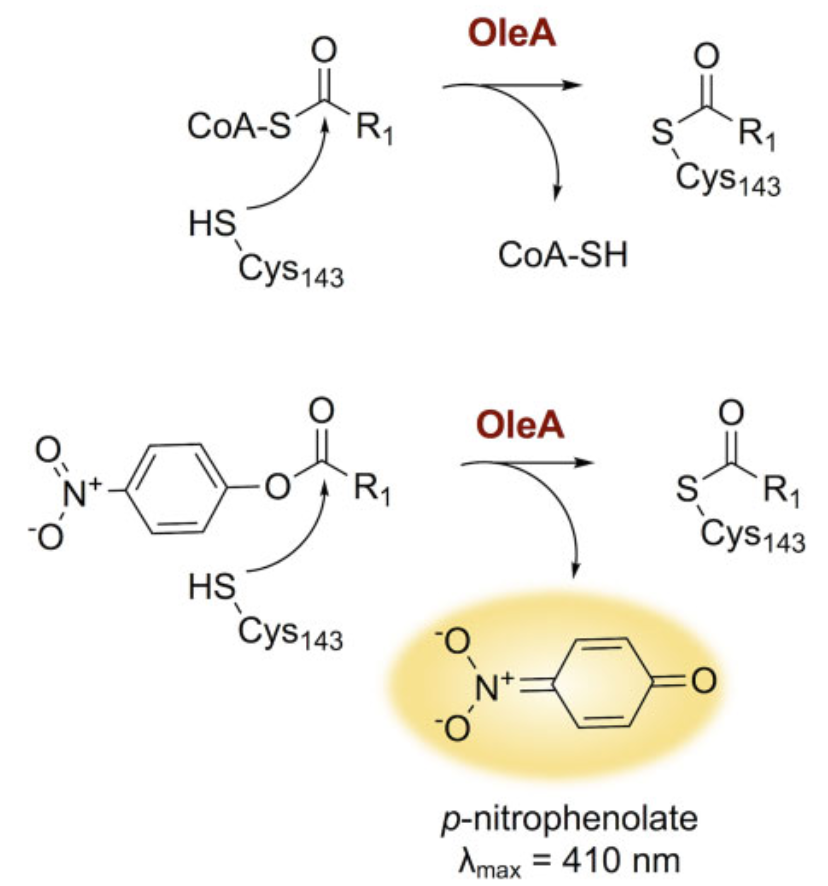
oleAの本来の基質はacyl-CoAです。このアシル基をoleAのCys143が脱離させます。この活性を調べる上で、p-nitrophenolateを用いた実験系が使われます(図1)。
結果
oleA類似配列59配列を対象にp-nitrophenolateの1種4-nitrophenyl-hexanoateを加水分解するか予測しました。
1.類似配列59配列の3次元構造予測
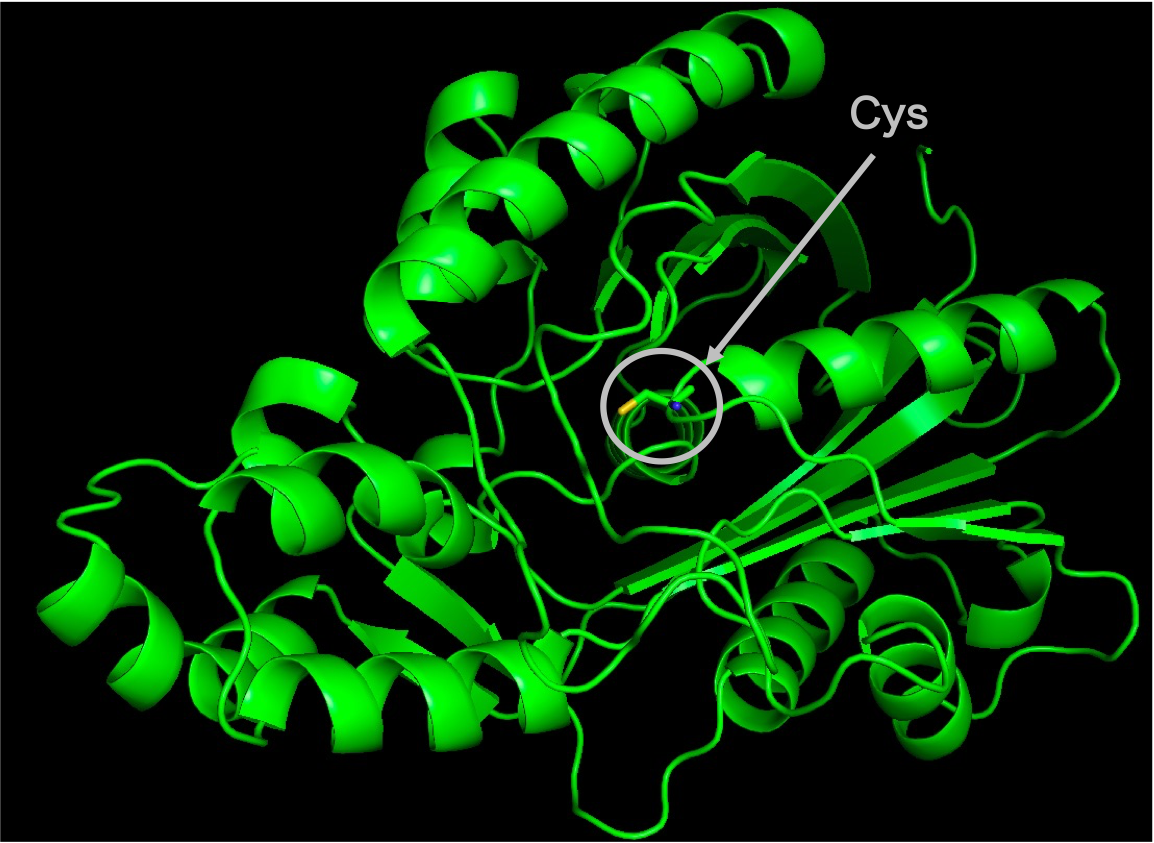
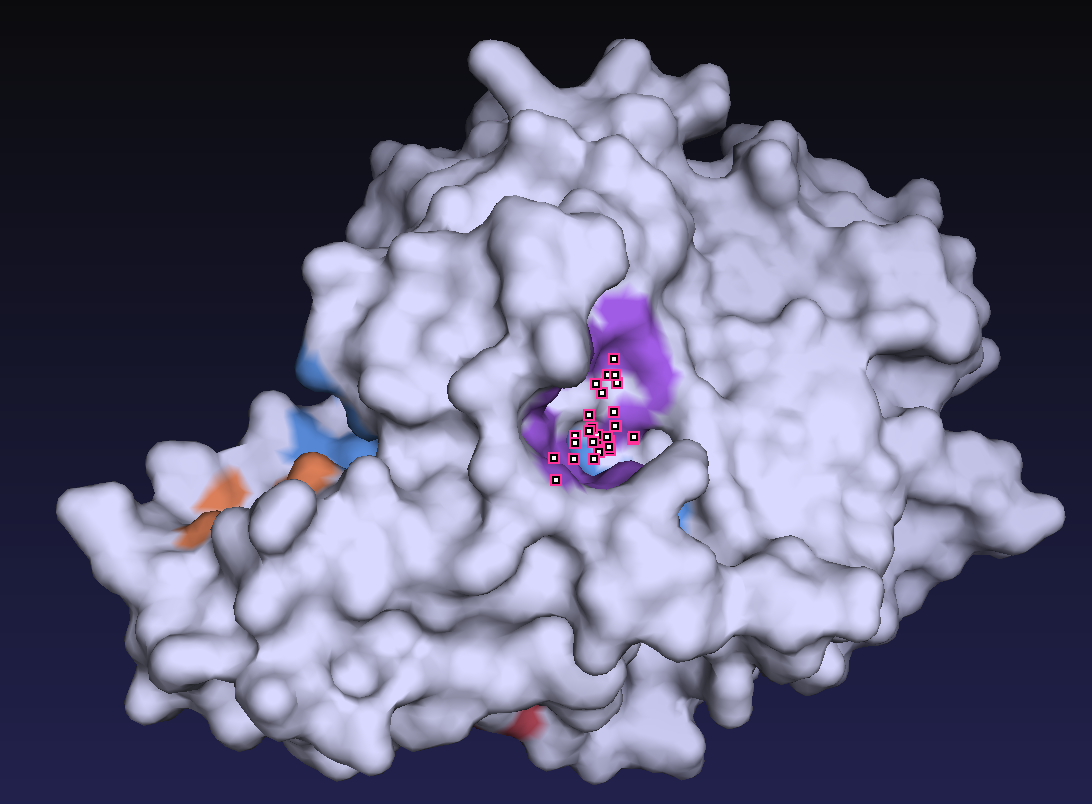
まずは類似配列59配列すべての構造が未知であるため、その3次元構造を予測し、予測した構造から活性残基の位置と基質が入るポケットの位置を予測しました。図2は類似配列の配列情報から予測した3次元構造および活性残基Cysの位置を示しています。図3が基質が結合するポケットの位置を予測した結果です。
2. 分子動力学シミュレーション
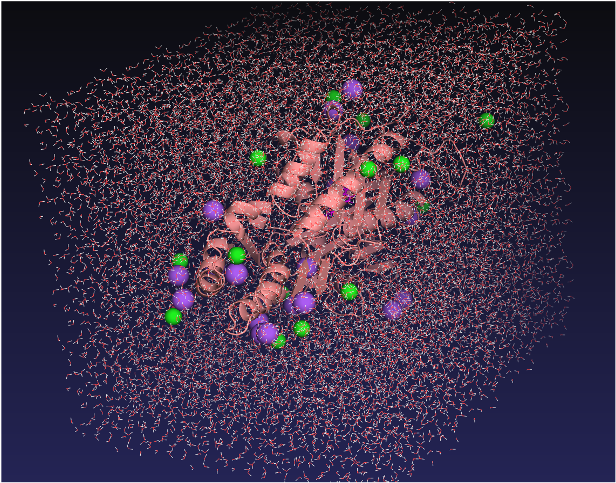
続いて、水分子とイオンの中に酵素と基質である4-nitrophenyl-hexanoateの複合体を配置して分子動力学シミュレーションを実行します(図4)。
3. digzyme独自酵素活性予測スコア算出
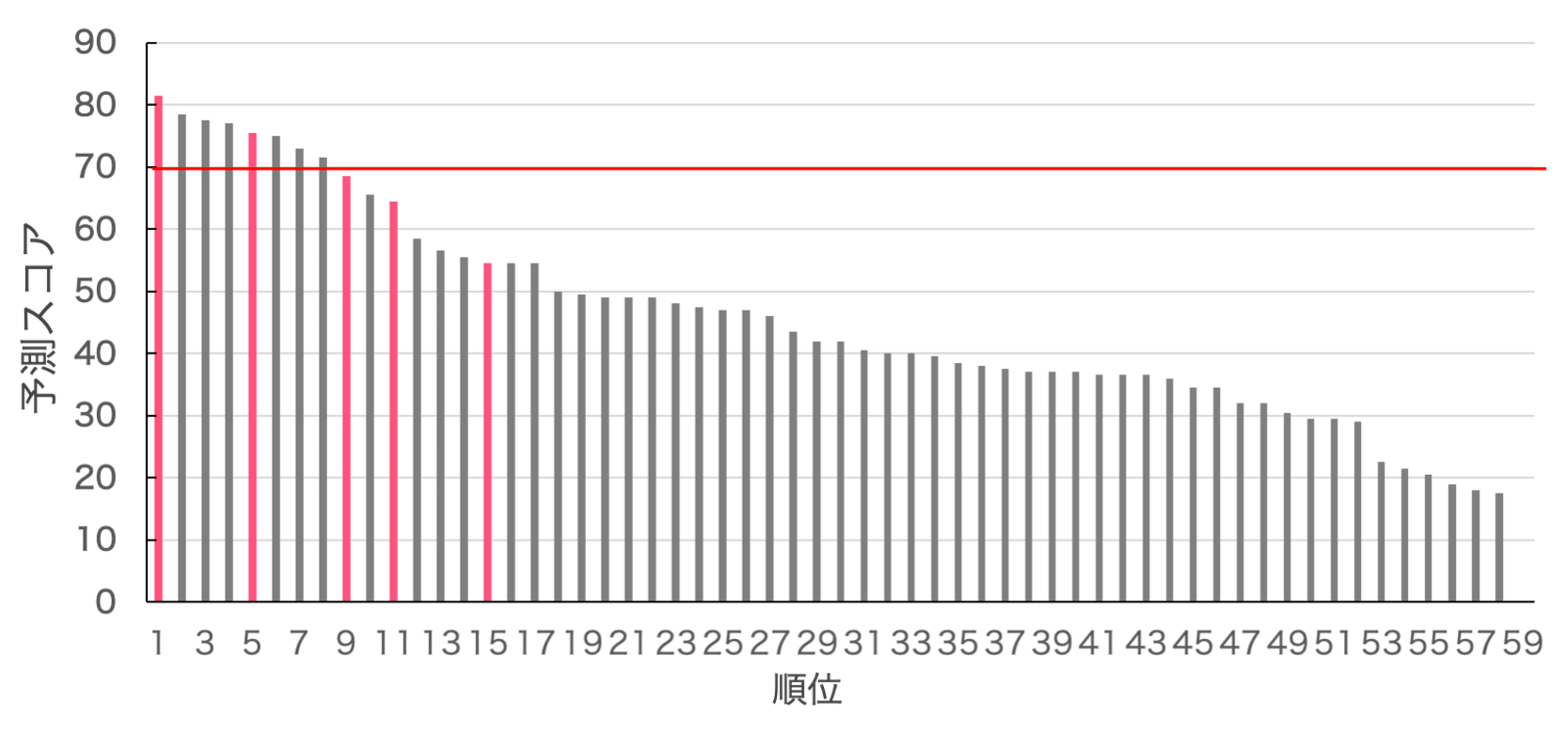
最後に分子動力学シミュレーションの結果から算出したdigzyme独自のスコアを計算します。図5にスコアが高い順に全59配列の予測スコアを記載しました。実証実験で活性があった配列をピンク色、活性がなかったものを灰色で表示しています。スコア70以上の配列を活性ありと判断しています(図5の赤線より上)。今回は9配列を活性ありと予測し、そのうち3配列が実際に活性を持っており、陽性適中率(PPV) = 0.30でした。また、真陽性率 (TPR) =0.6、偽陰性率 (FPR) =0.13という結果でした。このことから、不活性なものをランキング下位に分類できており、上位の配列に実際に活性のある配列を含んでいることが確認できました。
終わりに
本ブログでは、酵素活性予測技術を用いて、実験で活性が確認された酵素の活性予測を実演しました。通常実証実験を行う場合5~10配列を合成します。今回は上位5配列の中に実験で活性があった2配列が含まれており、弊社の酵素活性予測の精度が実用に適うものであると示されました。特に母集団の酵素配列のうち活性のあるものがわずかしか含まれていないようなケースを想定してデータを選びました(今回は59配列中5配列)。偽陰性率が低く抑えられているため、正しく酵素活性が予測できています。
謝辞
今回の酵素活性予測の材料として以下の論文から実証実験データを利用させていただきました。
Robinson et al., (2020) Machine learning-based prediction of activity and substrate specificity for OleA enzymes in the thiolase superfamily. Synthetic Biology.